Trang web cho phép download miễn phí bộ dữ liệu về cấu trúc các loại DNA và protein-Nguyễn Công Trình
Hôm nay mình xin chia sẻ cho cộng đồng biomdical data một trang web cho phép download miễn phí bộ dữ liệu về cấu trúc các loại DNA và protein, qua đó sẽ phục vụ các bạn trong quá trình nghiên cứu liên quan tới lĩnh vực này. Đó là trang Protein Data Bank.Tại trang này các bạn có thể tìm kiếm theo organism, structure feature ( macromolecule type, protein stoichiometry, enzym classfication.... Trang này cho phép ta visualize 3D cấu trúc của các đoạn DNA/protein đó. Các định dạng cho phép download rất đa dạng như: pdb, structure factor CIF, map coeffcient MTZ...2 hình dưới là một ví dụ về việc mình load cấu trúc một đoạn tetranucleosome vào công cụ mô phỏng Geant4-DNA để tính double strand break, single strand break, cluster strand break của đoạn DNA đó khi bị tương tác bởi một chùm proton.Link trang web: http://www.rcsb.org/
Tài nguyên này được cung cấp bởi kho lưu trữ Protein Data Bank - thông tin về hình dạng 3D của protein, axit nucleic và các tổ hợp phức tạp giúp sinh viên và nhà nghiên cứu hiểu tất cả các khía cạnh của y sinh và nông nghiệp, từ tổng hợp protein đến sức khỏe và bệnh tật.Là thành viên của wwPDB, RCSB PDB giám tuyển và chú thích dữ liệu PDB.
RCSB PDB xây dựng dựa trên dữ liệu bằng cách tạo ra các công cụ và tài nguyên cho nghiên cứu và giáo dục về sinh học phân tử, sinh học cấu trúc, sinh học tính toán, và hơn thế nữa.
pdb_extract là một tài nguyên tập hợp các chi tiết cụ thể về mô hình thử nghiệm và mô hình thử nghiệm của bạn từ các tệp đầu ra xác định cấu trúc và xác định cấu trúc của bạn để chuẩn bị cho việc lắng đọng PDB. Sử dụng công cụ trực tuyến này ( hướng dẫn có sẵn) hoặc tải xuống chương trình độc lập để chạy trên máy cục bộ của bạn. Công cụ này sẽ:
- cung cấp một biểu mẫu thông tin tác giả cho Xray, NMR , EM , có thể được lưu / cập nhật cho nhiều lắng đọng mục nhập liên quan.
- lắp ráp các tệp tọa độ và nhật ký liên quan đến các phương pháp thử nghiệm cụ thể của bạn.
- cho phép bạn sửa chữa chuỗi chính của chuỗi protein / nucleotide của bạn để giải quyết các dư lượng chưa được giải quyết.
===========
================CATEGORY 4: Authors of Structure============================ Enter authors of the deposited structures (at least one author) <structure_author_name= > !(e.g. Surname, F.M.) <structure_author_name= > <structure_author_name= > <structure_author_name= > <structure_author_name= > ...add more name if needed... ================CATEGORY 5a: Primary Citation ============================ The primary citation is the article in which the deposited coordinates were first reported. If the citation has not yet been published, give 'To be published' to the item 'primary_citation_journal_abbrev' and leave pages, year, volume blank. Enter the author name of primary citation <primary_citation_author_name= > !(e.g. Surname, F.M.) <primary_citation_author_name= > <primary_citation_author_name= > <primary_citation_author_name= > ...add more name if needed... Enter journal information of the primary citation <primary_citation_id= primary> <primary_citation_journal_abbrev= > (e.g. To be published) <primary_citation_title= > <primary_citation_year= > <primary_citation_journal_volume= > <primary_citation_page_first= > <primary_citation_page_last= > ================CATEGORY 5b: other citations (optional) ================ Other related citations may also be provided, if applicable. 1.Enter the author name of other citations <citation_author_id= > (e.g. 1, 2 ..) <citation_author_name= > <citation_author_name= > <citation_author_name= > <citation_author_name= > ...add more name if needed... 1. Enter journal information of the other citation <citation_id= 1 > (e.g. 1, 2, 3 ...) <citation_journal_abbrev= > <citation_title= > <citation_year= > <citation_journal_volume= > <citation_page_first= > <citation_page_last= > ...(add more other citations if needed)...
===================
Khái niệm cơ bản về cú pháp và định dạng
Định dạng PDBx / mmCIF sử dụng bộ ký tự ASCII.
Tất cả các mục dữ liệu được xác định theo tên, bắt đầu bằng ký tự gạch dưới và bao gồm tên danh mục và tên thuộc tính. Tên danh mục được phân tách khỏi tên thuộc tính theo một khoảng thời gian:
_citation.year
Sự kết hợp giữa danh mục và thuộc tính này có thể được gọi là mã thông báo mmCIF.
Các loại dữ liệu được trình bày theo hai kiểu: khóa-giá trị và dạng bảng.
Trong kiểu khóa-giá trị, mã thông báo mmCIF được theo dõi trực tiếp bởi một giá trị tương ứng. Ví dụ sau đây cho thấy các tham số ô đơn vị từ mục 4hhb:
_cell.entry_id 4HHB
_cell.length_a 63.150
_cell.length_b 83.590
_cell.length_c 53.800
_cell.angle_alpha 90.00
_cell.angle_beta
99.34
_cell.angle_gamma
_cell.length_a 63.150
_cell.length_b 83.590
_cell.length_c 53.800
_cell.angle_alpha 90.00
_cell.angle_beta
99.34
_cell.angle_gamma
Kiểu bảng được sử dụng khi có nhiều giá trị cho mỗi mã thông báo. Theo kiểu này, mã thông báo loop_ được theo sau bởi các hàng tên mục dữ liệu và sau đó là các giá trị dữ liệu được phân tách bằng khoảng trắng. Ví dụ sau đây cho thấy sự bắt đầu của các bản ghi tọa độ từ mục 4hhb. Ở đây, các mục dữ liệu trong danh mục Atom_site được sử dụng để mô tả danh tính và tọa độ nguyên tử của các nguyên tử trong mục:
loop_
_atom_site.group_PDB
_atom_site.id
_atom_site.type_symbol
_atom_site.label_atom_id
_atom_site.label_alt_id
_atom_site.label_comp_id
_atom_site.label_asym_id
_atom_site.label_entity_id
_atom_site.label_seq_id
_atom_site.pdbx_PDB_ins_code
_atom_site.Cartn_x
_atom_site.Cartn_y
_atom_site.Cartn_z
_atom_site.occupancy
_atom_site.B_iso_or_equiv
_atom_site.pdbx_formal_charge
_atom_site .auth_seq_id
_atom_site.auth_comp_id
_atom_site.auth_asym_id
_atom_site.auth_atom_id
_atom_site.pdbx_PDB_model_num
ATOM 1 N N. GIÁ TRỊ 1 1? 6.204 16.869 4.854 1.00 49.05? 1 GIÁ TRỊ 1
ATOM 2 C CA. GIÁ TRỊ 1 1? 6,913 17,759 4,607 1,00 43,14? 1 VAL A CA 1
ATOM 3 C C. GIÁ TRỊ 1 1? 8.504 17.378 4.797 1.00 24.80? 1 VAL AC 1
ATOM 4 O O. GIÁ TRỊ 1 1? 8.805 17.011 5.943 1.00 37,68? 1 GIÁ TRỊ AO 1
ATOM 5 C CB. GIÁ TRỊ 1 1? 6.369 19.044 5.810 1.00 72.12? 1 VAL A CB 1
ATOM 6 C CG1. GIÁ TRỊ 1 1? 7.009 20.127 5.418 1.00 61.79? 1 GIÁ TRỊ CG1 1
ATOM 7 C CG2. GIÁ TRỊ 1 1? 5.246 18.533 5.681 1.00 80.12? 1 GIÁ TRỊ CG2 1
_atom_site.group_PDB
_atom_site.id
_atom_site.type_symbol
_atom_site.label_atom_id
_atom_site.label_alt_id
_atom_site.label_comp_id
_atom_site.label_asym_id
_atom_site.label_entity_id
_atom_site.label_seq_id
_atom_site.pdbx_PDB_ins_code
_atom_site.Cartn_x
_atom_site.Cartn_y
_atom_site.Cartn_z
_atom_site.occupancy
_atom_site.B_iso_or_equiv
_atom_site.pdbx_formal_charge
_atom_site .auth_seq_id
_atom_site.auth_comp_id
_atom_site.auth_asym_id
_atom_site.auth_atom_id
_atom_site.pdbx_PDB_model_num
ATOM 1 N N. GIÁ TRỊ 1 1? 6.204 16.869 4.854 1.00 49.05? 1 GIÁ TRỊ 1
ATOM 2 C CA. GIÁ TRỊ 1 1? 6,913 17,759 4,607 1,00 43,14? 1 VAL A CA 1
ATOM 3 C C. GIÁ TRỊ 1 1? 8.504 17.378 4.797 1.00 24.80? 1 VAL AC 1
ATOM 4 O O. GIÁ TRỊ 1 1? 8.805 17.011 5.943 1.00 37,68? 1 GIÁ TRỊ AO 1
ATOM 5 C CB. GIÁ TRỊ 1 1? 6.369 19.044 5.810 1.00 72.12? 1 VAL A CB 1
ATOM 6 C CG1. GIÁ TRỊ 1 1? 7.009 20.127 5.418 1.00 61.79? 1 GIÁ TRỊ CG1 1
ATOM 7 C CG2. GIÁ TRỊ 1 1? 5.246 18.533 5.681 1.00 80.12? 1 GIÁ TRỊ CG2 1
Tên mục dữ liệu đầu tiên tương ứng với giá trị dữ liệu đầu tiên, mục thứ hai cho mục tiếp theo, v.v. trong mỗi dòng dữ liệu. Ví dụ: mục dữ liệu thứ ba _atom_site.type_symbol tương ứng với loại nguyên tử được đưa ra trong cột 13. Danh sách các mục dữ liệu, sau đó được lặp đi lặp lại thông qua các dòng dữ liệu.
Định dạng ví dụ
Một trong những lợi ích chính của định dạng PDBx / mmCIF là nó không đặt ra giới hạn nào cho số lượng nguyên tử, dư lượng hoặc chuỗi có thể được biểu diễn trong một mục nhập PDB
Ví dụ: bản ghi PDB có chứa các tác giả
AUTHOR G.FERMI,M.F.PERUTZ
Được biểu thị bằng PDBx / mmCIF với các mục dữ liệu trong danh mục aud_master:
loop_
_audit_author.name
_audit_author.pdbx_ordinal
'Fermi, G.' 1
'Perutz, M.F.' 2
_audit_author.pdbx_ordinal
'Fermi, G.' 1
'Perutz, M.F.' 2
Các thực thể
Một khái niệm mà định dạng PDBx / mmCIF dựa trên các thực thể. Một thực thể là một phần khác biệt về mặt hóa học của một cấu trúc như được biểu thị trong tệp dữ liệu PDBx / mmCIF. Các mục dữ liệu trong danh mục _entity, mô tả hóa học và danh tính của các phân tử đang được điều tra. Trong bất kỳ mục cụ thể nào, có thể có nhiều bản sao của một thực thể nhất định.
Ví dụ, cấu trúc 4hhb chứa hai bản sao của chuỗi alpha huyết sắc tố (hoặc chuỗi A và C) và hai bản sao của chuỗi beta (hoặc chuỗi B và D). Mục này cũng chứa bốn nhóm heme. Trong tệp PDBx / mmCIF, hai chuỗi alpha được coi là một thực thể, hai chuỗi beta là một thực thể khác và các nhóm heme là một phần ba. Các ion nước và phốt phát tạo thành các thực thể thứ tư và thứ năm:
Loop_
_entity.id
_entity.type
_entity.src_method
_entity.pdbx_description
_entity.formula_weight
_entity.pdbx_number_of_molecules
_entity.pdbx_ec
_entity.pdbx_mutation
_entity.pdbx_fragment
_entity.details
1 polymer man 'HEMOGLOBIN (DEOXY) (ALPHA CHAIN)' 15150.353 2 ? ? ? ?
2 polymer man 'HEMOGLOBIN (DEOXY) (BETA CHAIN)' 15890.198 2 ? ? ? ?
_entity.id
_entity.type
_entity.src_method
_entity.pdbx_description
_entity.formula_weight
_entity.pdbx_number_of_molecules
_entity.pdbx_ec
_entity.pdbx_mutation
_entity.pdbx_fragment
_entity.details
1 polymer man 'HEMOGLOBIN (DEOXY) (ALPHA CHAIN)' 15150.353 2 ? ? ? ?
2 polymer man 'HEMOGLOBIN (DEOXY) (BETA CHAIN)' 15890.198 2 ? ? ? ?
3 non-polymer syn 'PROTOPORPHYRIN IX CONTAINING FE' 616.487 4 ? ? ? ?
4 non-polymer syn 'PHOSPHATE ION' 94.971 2 ? ? ? ?
4 non-polymer syn 'PHOSPHATE ION' 94.971 2 ? ? ? ?
5 water nat water 18.015 221 ? ? ? ?
Các phiên bản gần đây của gói sàng lọc Phenix, REFMAC và Buster tạo các tệp PDBx / mmCIF sẵn sàng để lắng đọng:
Phenix: Hướng dẫn có sẵn tại trang web của Phenix, https://www.phenix-online.org/documentation/overview/xray-structure-depocation.html
CCP4: hướng dẫn có sẵn cho CCP4i2 ( http://www.ccp4.ac.uk/depocation_ccp4i2 ) hoặc CCP4 Cloud ( http://www.ccp4.ac.uk/depocation_ccp4cloud )
REFMAC (khi sử dụng bên ngoài Đám mây CCP4i2 hoặc CCP4): Để xuất tệp PDBx / mmCIF từ REFMAC, hãy thêm thẻ đọc "định dạng pdbout mmcif". REFMAC cũng có thể đọc tệp bằng cách chỉ định tệp PDBx / mmCIF làm đối số HKLINE.
=================
=================
Vietnamen’s Weblog
Time, Chances, Diligence, Intelligence: which is the most important?
Posts Tagged ‘protein’
Protein resources
leave a comment »
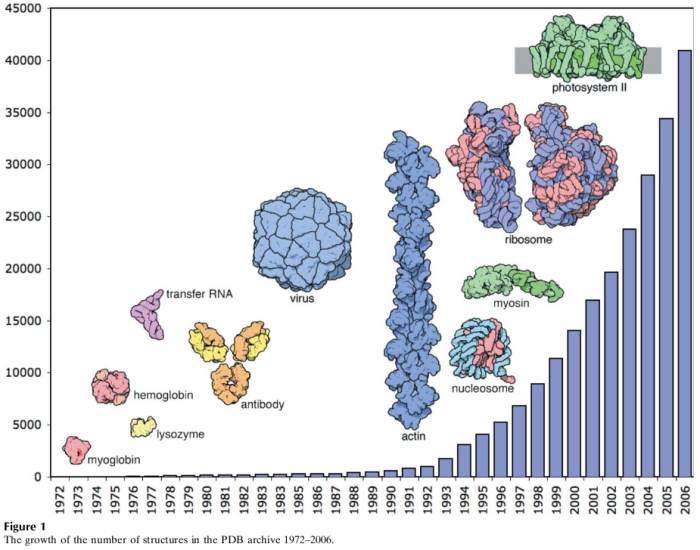
Lịch sửcủa quá trình phát hiện cấu trúc protein (Berman, 2008).

- http://journals.iucr.org/a/issues/2008/01/00/sc5004/sc5004.pdf
- http://nar.oxfordjournals.org/cgi/content/full/28/1/235 (The Protein Data Bank)
Protein Data Bank (PDB)
– Cấu trúc 3D của các proteins được lưu trữ dưới dạng text file – qua 2 formats chính là PDB (column-based) và PDBML (XML-based file format)
– Cấu trúc 3D của mỗi protein được xác định bằng thực nghiệm nhờ vào các kĩ thuật phức tạp là X-ray crystallography và proteinNMR.
– Các nhà nghiên cứu muốn công bố cấu trúc của một protein mà họ tìm ra phải cung cấp thông tin theo chuẩn PDB. Vì thế việc truy xuất chúng khá dễ dàng nhờ Internet.
Links:
- http://www.pdb.org/pdb/home/home.do (Truy xuất thông tin PDB của proteins nếu biết ID của nó hoặc tên tác giả công bố)
- http://www.rcsb.org/robohelp_f/#structure_explorer/summary_information.htm (Cấu trúc file PDB)
- http://www.rcsb.org/pdb/static.do?p=education_discussion/Looking-at-Structures/coordinates.html
Molecular Visualization Software
– Cấu trúc 3D được lưu trữ trong file PDB có thể được tái tạo lại bằng các công cụ đồ họa như VMD, PyMOL…
- http://www.umass.edu/microbio/rasmol/othersof.htm
- http://www.molvis.indiana.edu/C571_F02/molvis_software.html
Molecular Dynamic Simulations
– Thông tin thực nghiệp thu thập được (lưu trong PDB) giúp ta có thể tính được tương tác giữa các atom trong một residue, giữa residue này và residue khác (ví dụ: covalent bond, non-covalent bond, bond stretch, angle stretch…) nhờ vào forcefield đã được thiết lập. Từ đó giúp tìm ra conformation của protein ở trạng thái cân bằng (minimized energy). Đồng thời quá trình biến đổi từ trạng thái ban đầu sang cân bằng có thể được minh họa bằng các dùng các tool trong Molecular Visualization Software. Việc tính toán là quá trình rất phức tạp, tốn thời gian và có các molecular dynamics softwares hỗ trợ (free lẫn commercial). Trong đó rất phổ biến là CHARMM.
Links:
- http://en.wikipedia.org/wiki/Category:Molecular_dynamics_software
- http://www.charmm.org/
- http://www.charmming.org/charmming/
- http://www.charmm-gui.org/
- http://en.wikipedia.org/wiki/Force_field_(chemistry)
- http://en.wikipedia.org/wiki/Category:Intermolecular_forces
- http://en.wikipedia.org/wiki/Water_model (TIP3, TIP4P…)
Nguồn : Hoàng Hà
